2.6 Stammbäume in R
Ein Stammbaum (auch Phylogenie, englisch phylogeny) ist ein Modell der evolutionären Beziehungen zwischen Abstammungslinien, zum Beispiel zwischen Arten. Phylogenien sind für das Verständnis der Evolution über lange Zeiträume von zentraler Bedeutung. Bevor wir uns damit befassen, wie man einen Stammbaum aus Daten wie DNA ableitet, geht es in dieser Übungseinheit darum, wie man Stammbäume in R einliest, interpretiert und visualisiert.
2.6.1 Lernziele
Nach dieser Übungseinheit könnt ihr:
einen Stammbaum mit R lesen, speichern und darstellen
Knoten, Spitzen, Kladen und Äste eines Stammbaums identifizieren
monophyletische, paraphyletische und polyphyletische Gruppen unterscheiden
2.6.2 Literatur
Liam Revell, Introduction to phylogenies in R
2.6.4 Tutorial
2.6.4.1 Die Elemente eines Stammbaums
Wir werden in dieser Einheit die Baobab-Gattung Adansonia (Malvaceae, Unterfamilie Bombacoideae) als Beispiel verwenden.
![]()
Die folgende Grafik zeigt den Stammbaum von Adansonia (aus Zizka et al. 2018):
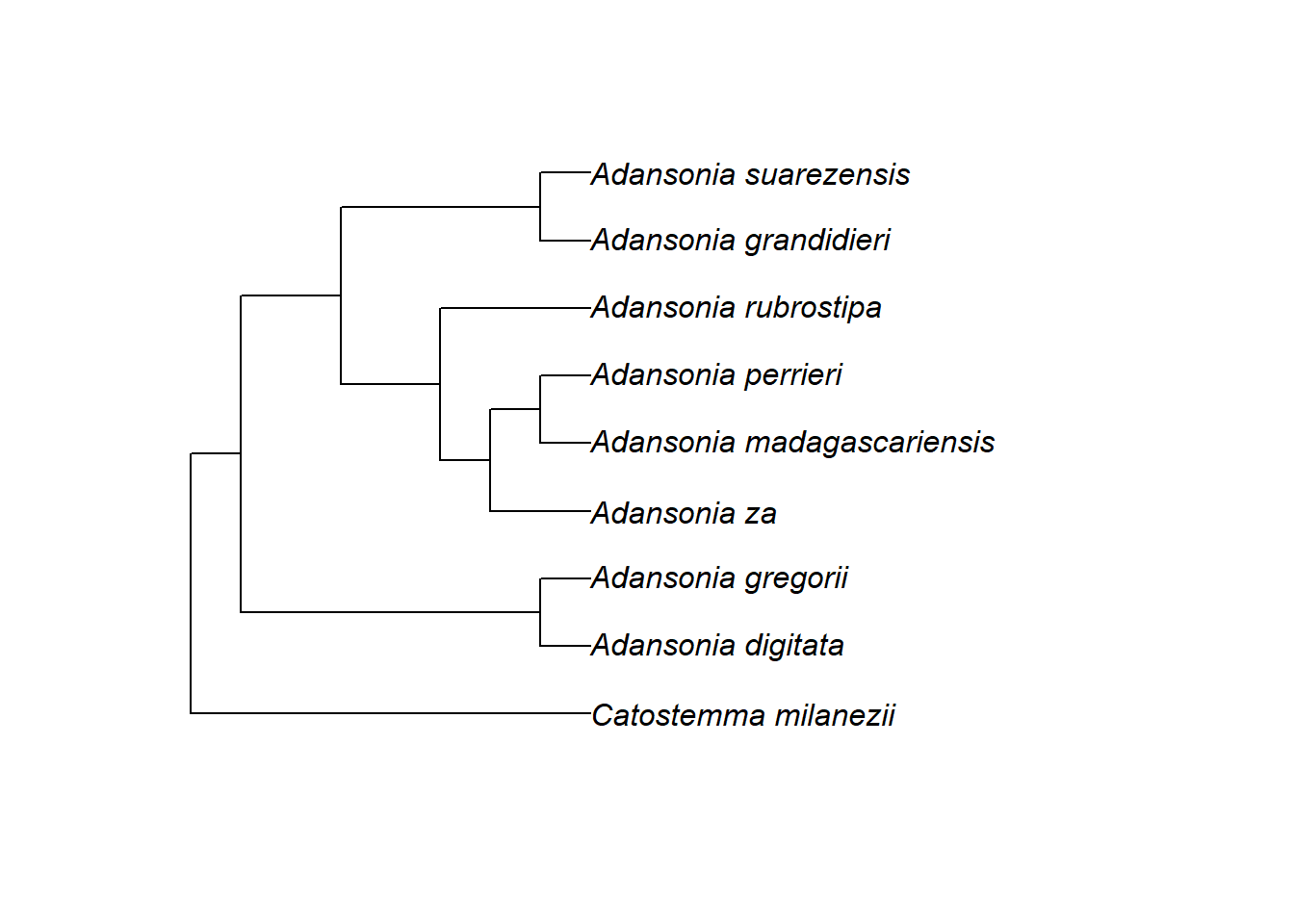
Man sieht die drei Grundelemente eines Stammbaums:
Die Spitzen (tips) oder Blätter: Dies sind die Endknoten, das heißt, sie haben keine weiteren Nachkommen. Hier entsprechen sie neun Arten.
Die (internen) Knoten (nodes): Das sind die Stellen, an denen sich zwei Arten in der Vergangenheit voneinander entfernt haben. Ihr seht, dass dieser Stammbaum dichotom ist, was bedeutet, dass jeder interne Knoten genau zwei Nachkommen hat.
Die Äste (branches oder edges): Sie verbinden die internen Knoten und die Endknoten. Sie entsprechen einer evolutionären Linie von Vorfahre zu Nachkommen.
Ihr seht auch, dass es neben den acht Arten von Adansonia noch eine Außengruppe gibt, Catostemma milanezii. Mehr zu Außengruppen in der nächsten Übungseinheit.
Wie kommt man nun mit R zu einem Diagramm wie dem oben gezeigten? Fangen wir von vorne an.
2.6.4.2 Setup
Bevor wir uns speziell mit Stammbäumen beschäftigen, müssen wir ein paar Dinge einrichten, damit wir arbeiten können. Dieses Setup nutzen wir auch für die folgenden Übungseinheiten.
Erstellt zunächst einen Kursordner mit den Unterverzeichnissen scripts, data und results in eurem Netzwerkverzeichnis, wie unter Übungseinheiten angegeben.
Speichert ein leeres Skript in RStudio in eurem Verzeichnis scripts unter dem Namen 01_stammbaum.R: Datei > Speichern unter.
Verlasst RStudio, sucht euer R-Skript und öffnet es. Euer PC sollte die .R-Endung erkennen und automatisch RStudio verwenden.
Euer Arbeitsverzeichnis sollte nun jenes sein, in dem ihr euer R-Skript geöffnet habt. Überprüft dies, indem ihr in der R-Konsole (unten links) Folgendes eingebt.
Ein wichtiges Paket für die Arbeit mit Stammbäumen in R ist ape (analysis of phylogenetics and evolution).
Installiert und ladet das Paket:
2.6.4.3 Stammbäume in R
Ladet die Datei adansonia.txt aus dem ILIAS Ordner in euer /data-Verzeichnis herunter. Öffnet sie in einem Texteditor (Notepad oder ähnlich). Sie sollte wie folgt aussehen:
## (Catostemma_milanezii,((Adansonia_digitata,Adansonia_gregorii),(((Adansonia_za,(Adansonia_madagascariensis,Adansonia_perrieri)),Adansonia_rubrostipa),(Adansonia_grandidieri,Adansonia_suarezensis))));Versucht, diese Textstruktur zu verstehen. Vergleicht sie mit dem Diagramm des Stammbaums am Anfang des Tutorials. Was bedeuten die Klammern, die Kommata und das Semikolon?
Lasst uns das gemeinsam an der Tafel analysieren.
Diese Textdarstellung ist das Newick-Format, das Standardformat für Stammbäume. Es wird von den meisten Phylogenetik-Programmen verwendet.
Lesen wir nun diesen Stammbaum in R ein und speichern ihn in einer Variablen namens tree:
# zwei Punkte (..) in dem Pfad zur Datei stehen für das Elternverzeichnis
tree <- read.tree("../data/adansonia.txt")tree ist ein Objekt der Klasse „phylo“:
## [1] "phylo"Wenn ihr nur den Namen des Objekts, tree, eingebt, erhaltet ihr eine Übersicht:
##
## Phylogenetic tree with 9 tips and 8 internal nodes.
##
## Tip labels:
## Catostemma_milanezii, Adansonia_digitata, Adansonia_gregorii, Adansonia_za, Adansonia_madagascariensis, Adansonia_perrieri, ...
##
## Rooted; no branch length.2.6.4.4 Plotten eines Stammbaums als Grafik
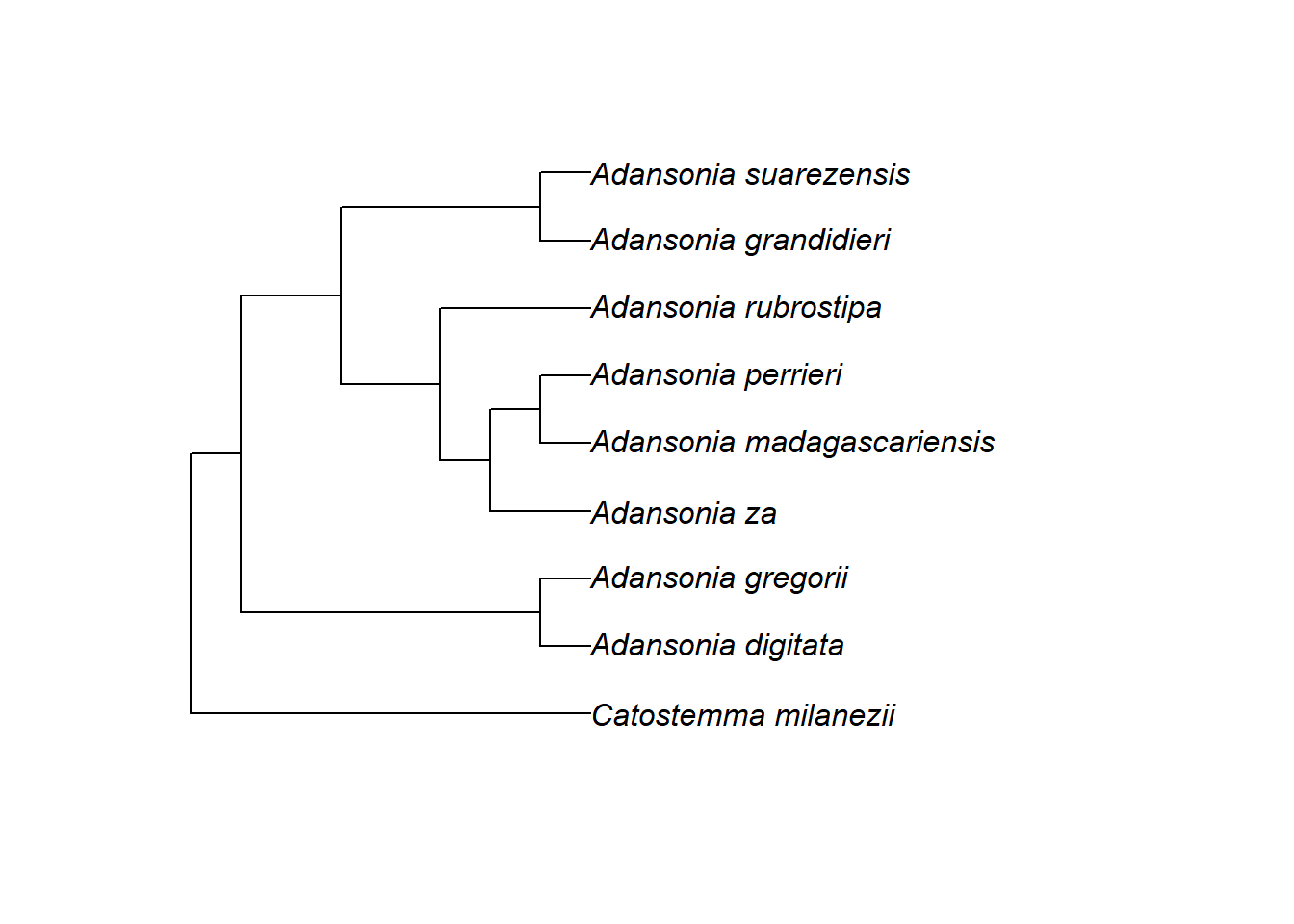
Da tree zur Objektklasse „phylo“ gehört, weiß R bereits, wie es zu interpretieren ist, um eine Grafik mit sinnvollen Standardparametern zu erstellen.
Erstelle eine einfache Darstellung von tree:
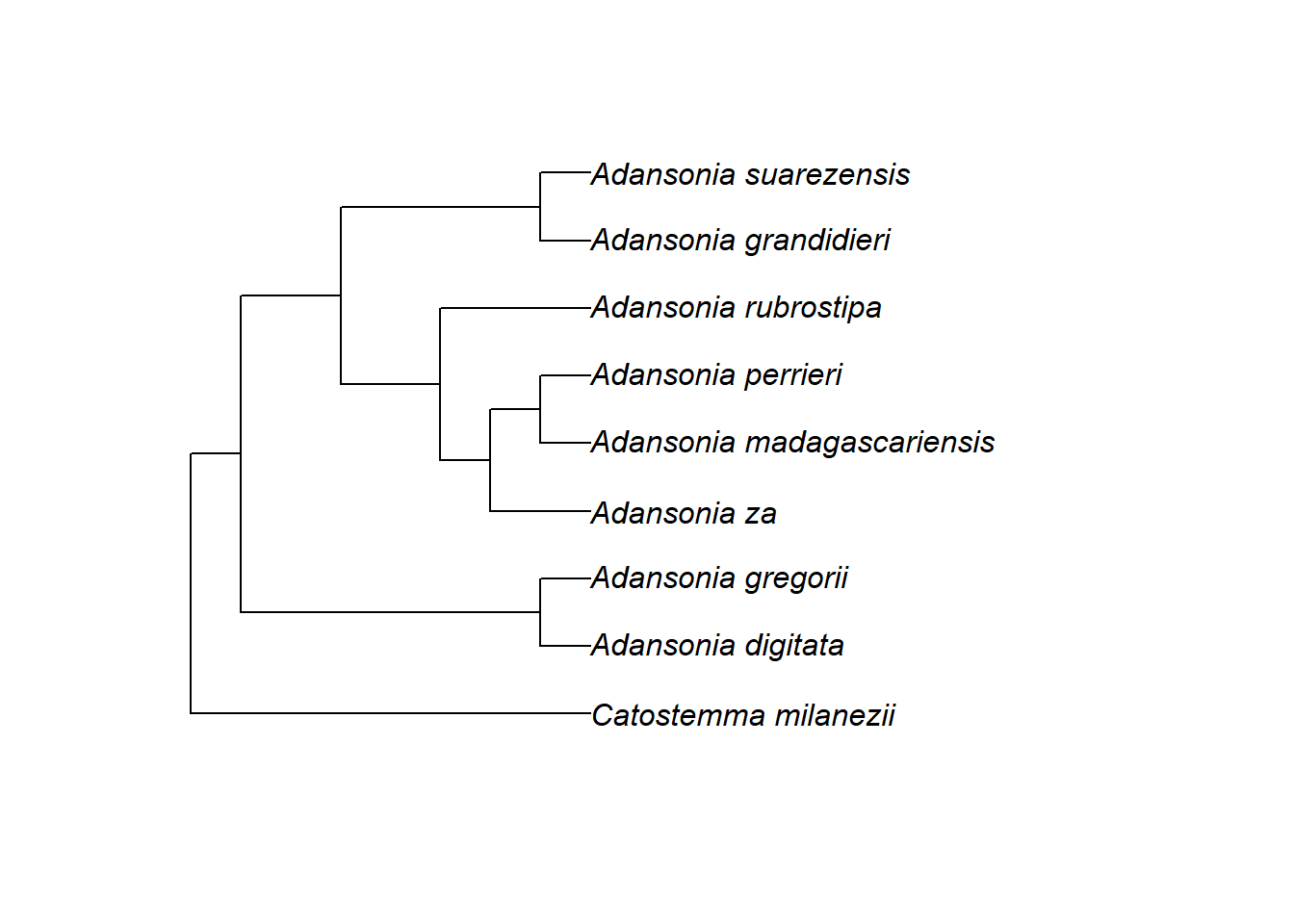
Durch Änderung der Argumente von plot.phylo() können wir die Grafikparameter anpassen, um z.B. die Farbe der Beschriftung zu ändern:
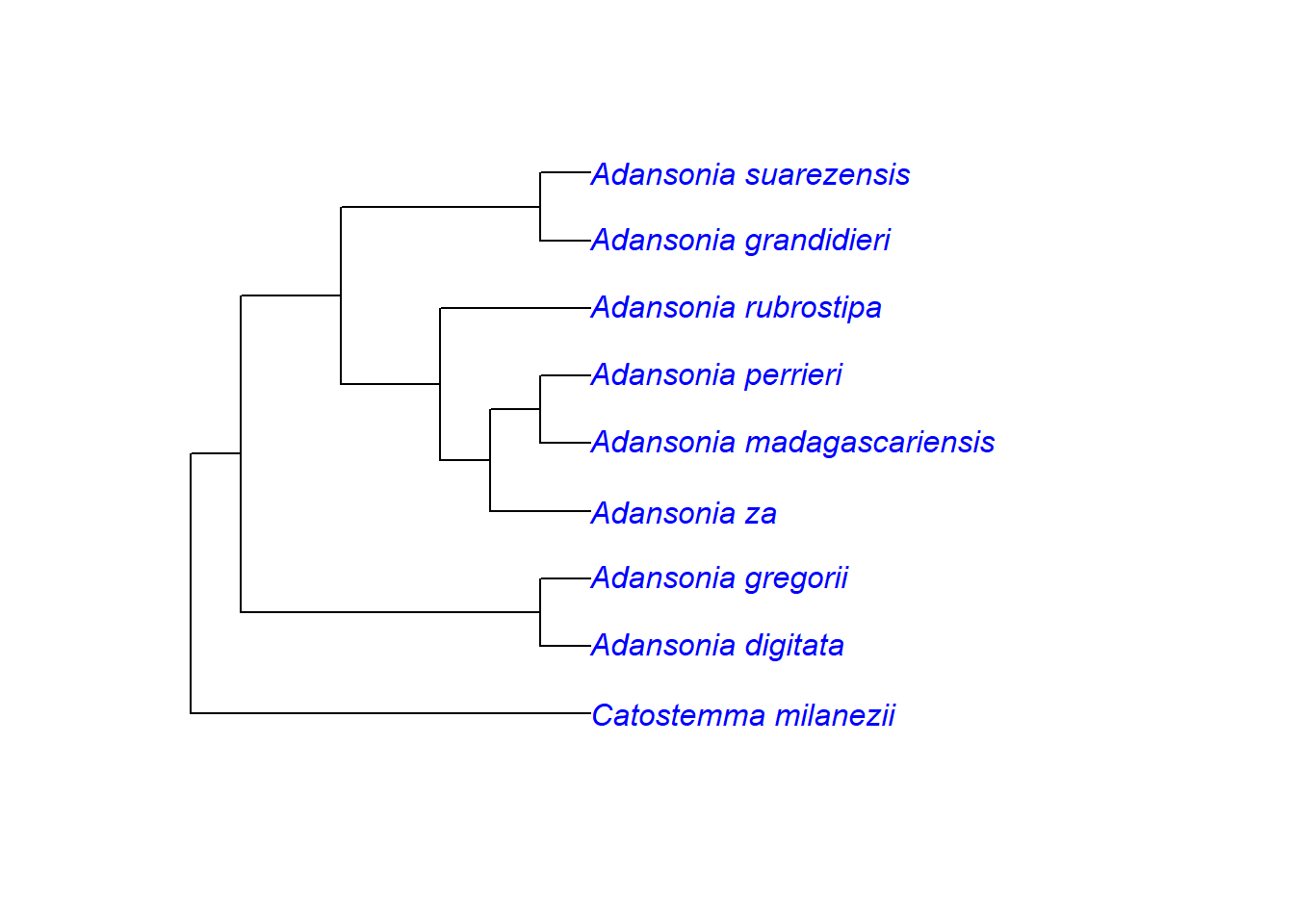
Es gibt verschiedene Möglichkeiten, einen Stammbaum darzustellen. Versucht es zum Beispiel mit einem Kladogramm. Es vermittelt dieselben Informationen, nur in einem anderen grafischen Stil:
2.6.4.5 Spitzen, Knoten und Äste
Versuchen wir nun zu verstehen, wie die drei grundlegenden Elemente eines Stammbaums in R dargestellt werden.
Ein phylo-Objekt ist ein spezieller Typ einer Liste (in R ein Objekt, das andere Objekte enthält). Wie viele und welche Objekte sind in tree enthalten?
## [1] 3## [1] "edge" "Nnode" "tip.label"Ein phylo-Objekt in R enthält immer diese drei Elemente. Ihr könnt sie jeweils mit dem Dollarzeichen $ auswählen oder aufrufen.
Die Namen der Spitzen (tips) – hier die Artnamen – werden als Zeichenvektor gespeichert:
## [1] "Catostemma_milanezii" "Adansonia_digitata"
## [3] "Adansonia_gregorii" "Adansonia_za"
## [5] "Adansonia_madagascariensis" "Adansonia_perrieri"
## [7] "Adansonia_rubrostipa" "Adansonia_grandidieri"
## [9] "Adansonia_suarezensis"Beachtet, dass diese Namen Unterstriche (_) enthalten, die Grafik jedoch nicht! Das liegt daran, dass die Funktion plot.phylo() standardmäßig keine Unterstriche darstellt. Leerzeichen können in der Newick-Datei zu Problemen führen, daher werden sie im Allgemeinen durch Unterstriche ersetzt.
Wir können leicht auf die Namen der Spitzen zugreifen und sie ändern. Kopieren wir zunächst den Stammbaum als neue Variable und ersetzen dann einen Namen – den zweiten – durch einen neuen.
## [1] "Catostemma_milanezii" "Adansonia_digitata"
## [3] "Adansonia_gregorii" "Adansonia_za"
## [5] "Adansonia_madagascariensis" "Adansonia_perrieri"
## [7] "Adansonia_rubrostipa" "Adansonia_grandidieri"
## [9] "Adansonia_suarezensis"# Da es sich bei diesen Werten um Zeichenketten und nicht um Zahlen handelt, müssen wir sie für R in Anführungszeichen setzen
tree2$tip.label[2] <- "A. digitata"
tree2$tip.label## [1] "Catostemma_milanezii" "A. digitata"
## [3] "Adansonia_gregorii" "Adansonia_za"
## [5] "Adansonia_madagascariensis" "Adansonia_perrieri"
## [7] "Adansonia_rubrostipa" "Adansonia_grandidieri"
## [9] "Adansonia_suarezensis"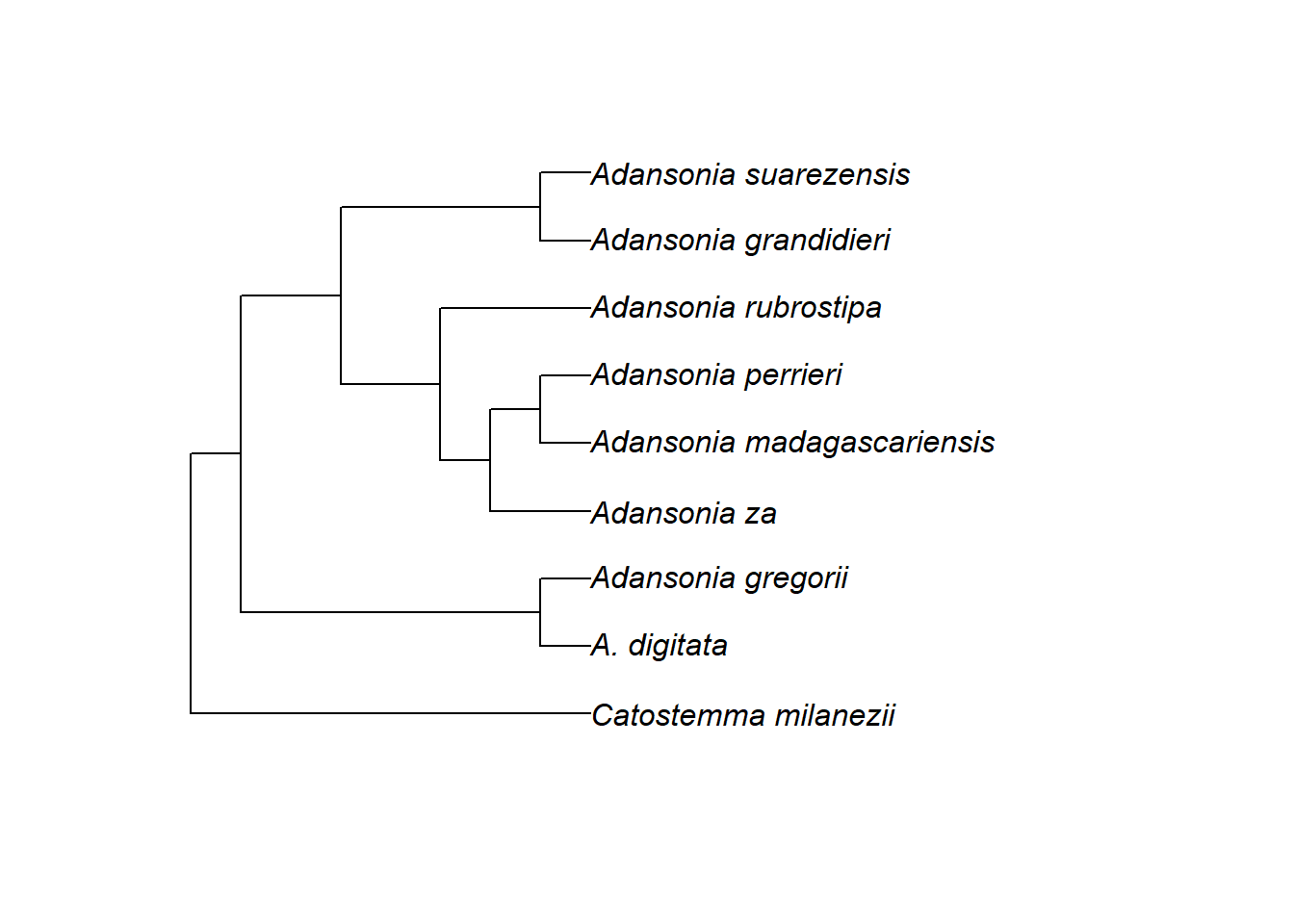
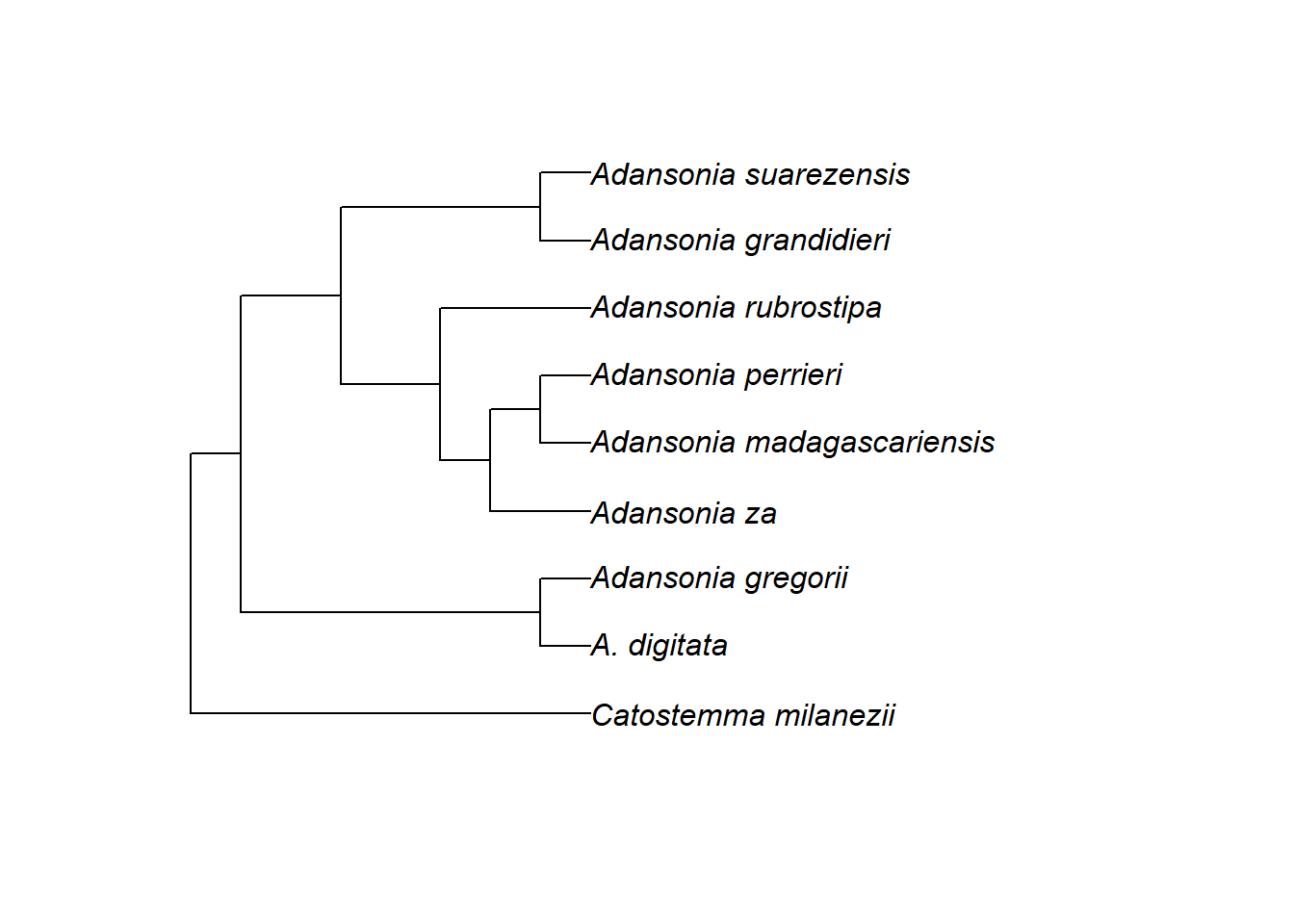
Schauen wir uns nun die Knoten an. In einem phylo-Objekt sind die Knoten nummeriert, womit wir bestimmte Knoten ansprechen können. Um alle Knotennummern in einem Baum anzuzeigen, können wir nodelabels() verwenden:
Dies ermöglicht es uns nun, einen bestimmten Knoten zu identifizieren, um zum Beispiel den Vorfahren von Adansonia suarezensis und A. grandidieri (Knoten Nr. 17) in der Grafik mit einem Punkt (pch = 20) zu kennzeichnen, der dreimal so groß ist wie die Standardgröße (cex = 3):
plot.phylo(tree)
# Beachtet, dass plot.phylo() immer eine neue Grafik beginnt, während nodelabels() einer bestehenden Grafik hinzugefügt wird
nodelabels(node = 17, pch = 20, cex = 3)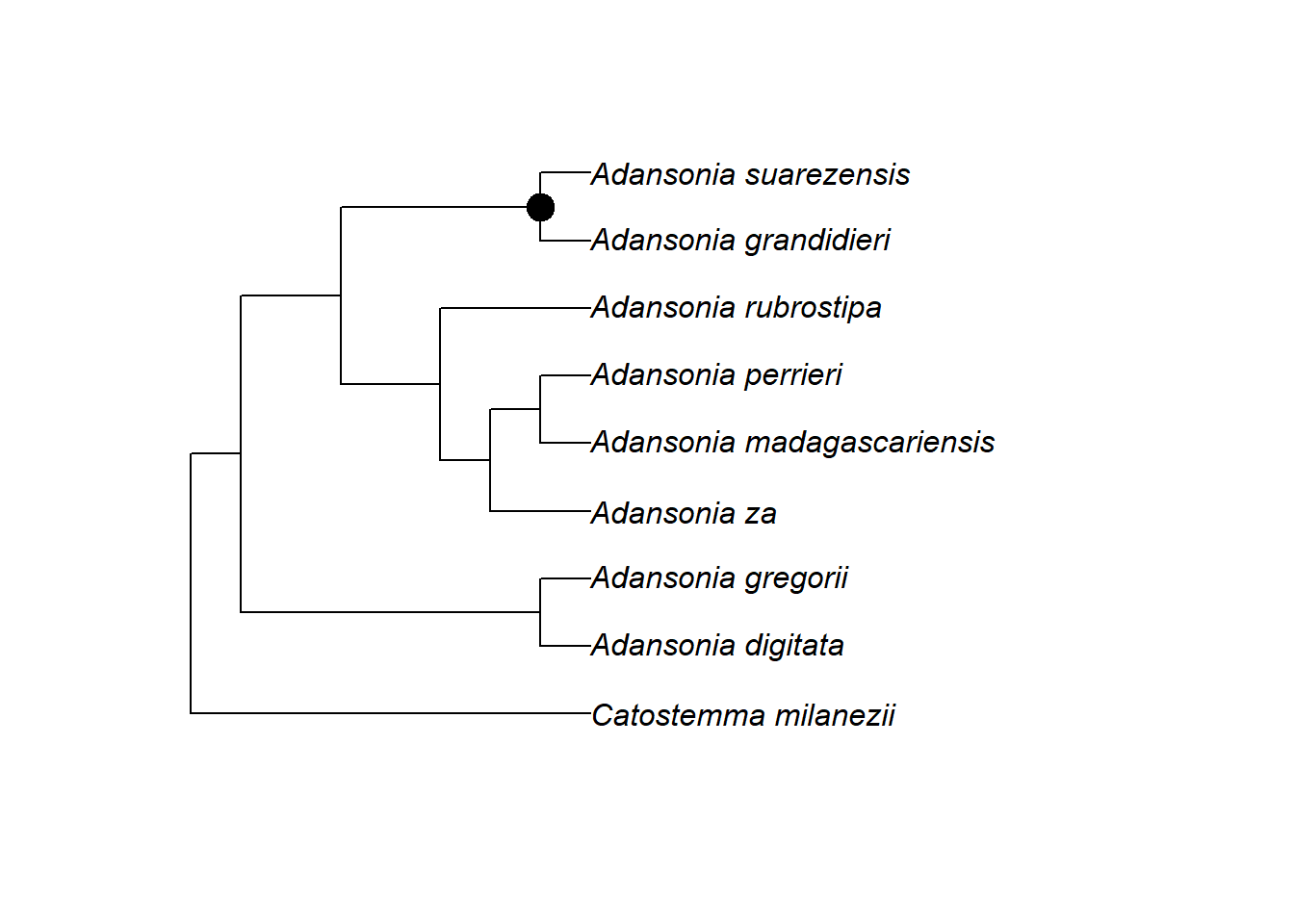
Ihr seht, dass die Nummern beim Knoten ganz links, der Wurzel, beginnen, aber nicht mit 1. Das liegt daran, dass die Spitzen auch Knoten sind, und die Nummerierung der Knoten in ape mit ihnen beginnt:
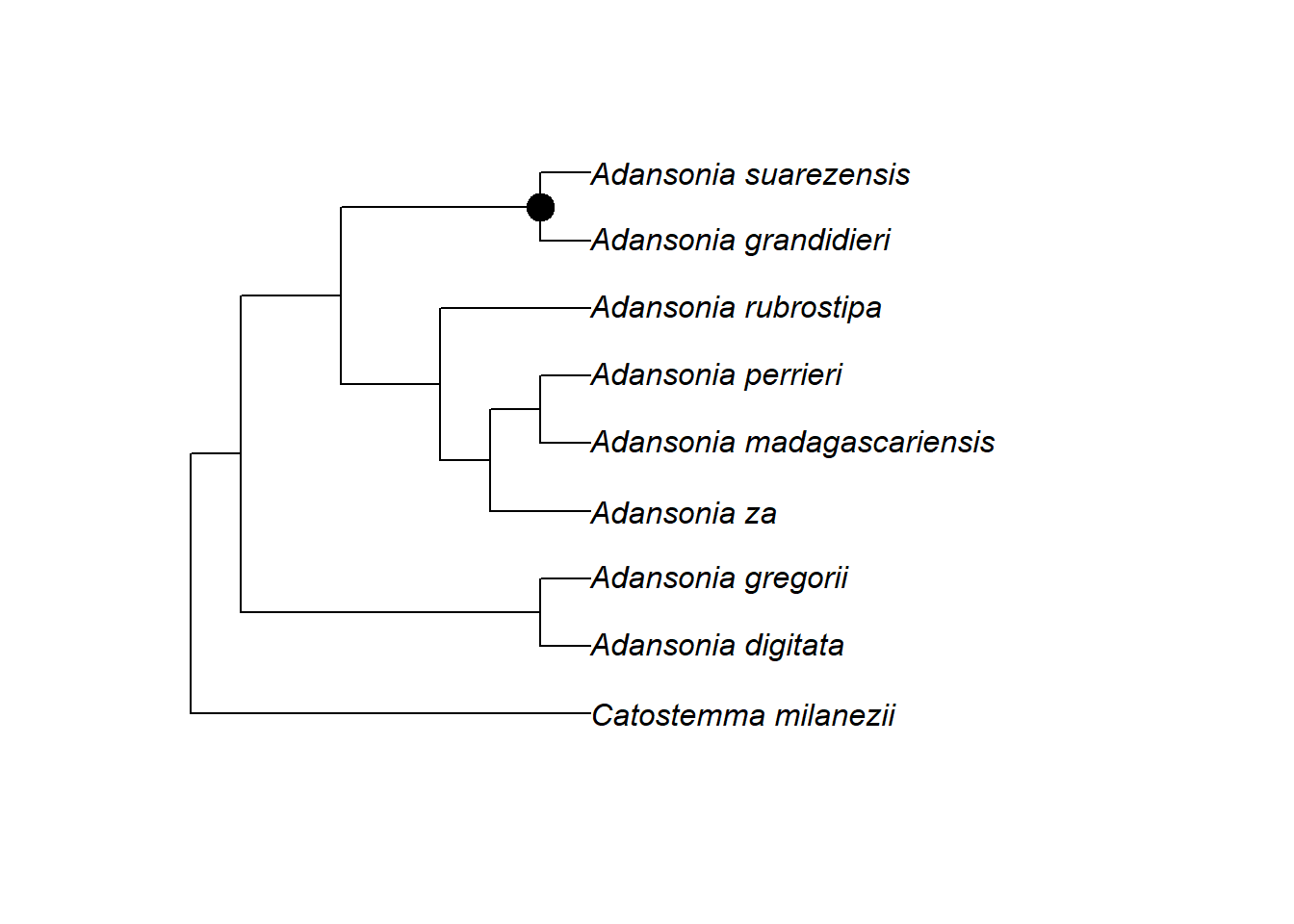
2.6.4.6 Kladen und Vorfahren
Aus der Vorlesung werdet ihr euch daran erinnern, dass eine Gruppe, die alle von einem Vorfahren abstammenden Spitzen vereint, als monophyletische Gruppe bezeichnet wird. Ein Synonym dafür ist Klade (clade).
Für jedes Paar oder jede Gruppe von Spitzen können wir ihren jüngsten gemeinsamen Vorfahren (most recent common ancestor, oft mit dem Akronym MRCA bezeichnet) identifizieren. Um zum Beispiel den MRCA der gesamten Gattung Adansonia zu bestimmen, brauchen wir nur zwei Spitzen:
# wir geben die Spitzen als Zeichenvektor an
getMRCA(phy=tree, tip=c("Adansonia_suarezensis", "Adansonia_digitata"))## [1] 11Die obige Funktion gibt die Knotennummer des MRCA aus. Wir können diese in einer Variablen n speichern und den Knoten in einer Grafik markieren, wie wir es oben getan haben:
n <- getMRCA(phy = tree, tip = c("Adansonia_suarezensis", "Adansonia_digitata"))
plot.phylo(tree)
nodelabels(node = n, pch = 20, cex = 3)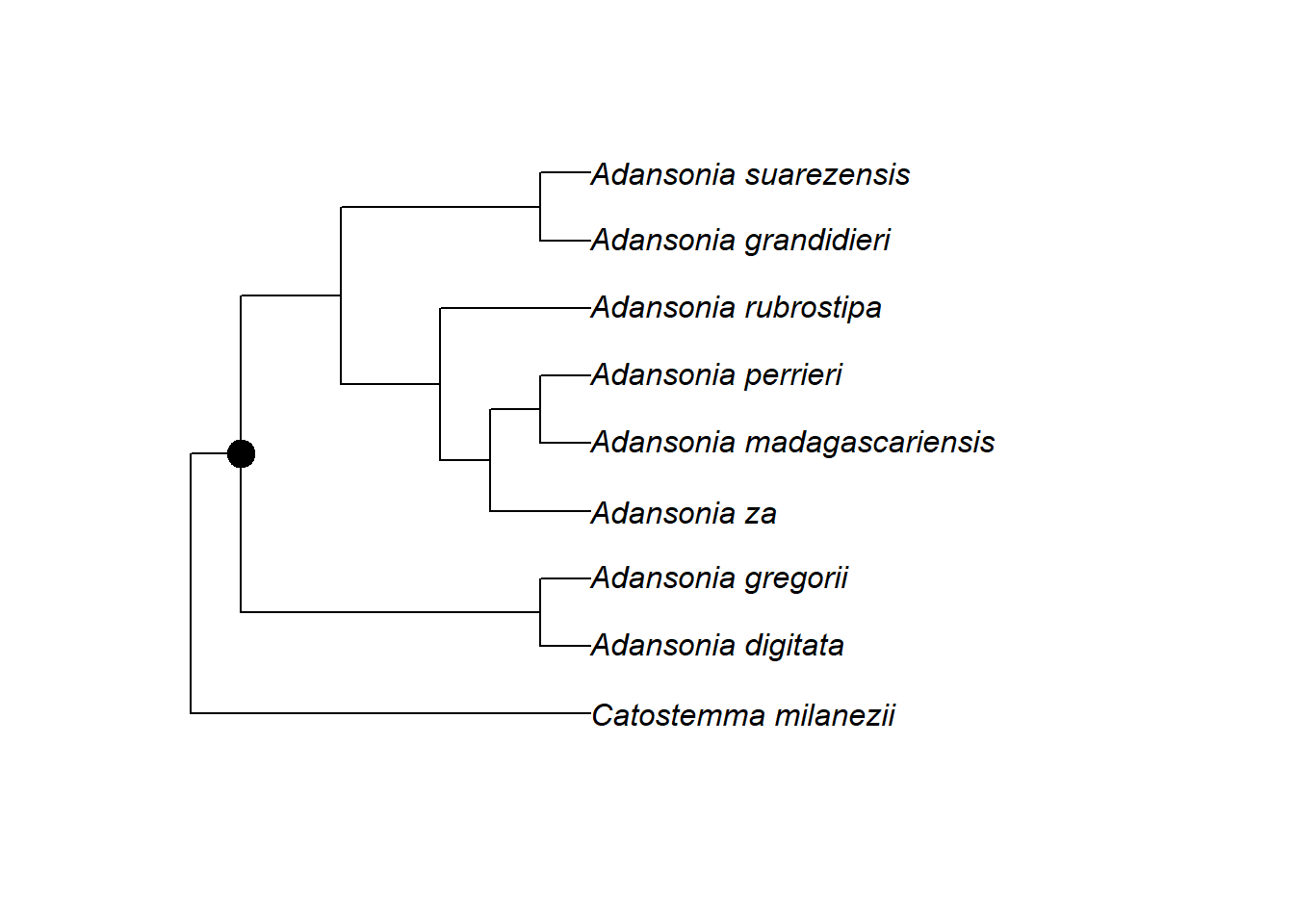
R kann uns auch sagen, ob eine Gruppe monophyletisch ist:
## [1] TRUE## [1] FALSEIhr seht, dass A. madagascariensis und A. za keine Klade oder monophyletische Gruppe bilden, da ihr MRCA einen weiteren Nachkommen hat, Adansonia perrieri. Die drei Arten zusammen sind monophyletisch, aber A. madagascariensis und A. za sind eine paraphyletische Gruppe.
2.6.4.7 Speichern eines Stammbaums
Das Gegenstück zu read.tree() ist write.tree(). Speichern wir z.B. den Baum tree2, in dem wir oben einen Namen geändert haben, im Ordner /results:
Die Dateiendung .nwk ist nur eine Konvention. Man sieht auch oft .tre, könnte aber auch etwa .txt verwenden – es ist einfach eine Textdatei. Wenn ihr sie in einem Texteditor öffnet, werdet ihr das Newick-Format wiedererkennen, das wir oben gesehen haben.
2.6.5 Aufgaben
- Schaut euch die verschiedenen Argumente für
plot.phylo()an, indem ihr die Funktionshilfe aufruft (?plot.phylo). Probiert andere Darstellungsarten aus. Wie kann man die Farbe der Äste, die Schriftgröße und die Schriftart ändern?
## starting httpd help server ... done


Ihr habt mono- und paraphyletische Gruppen gesehen. Wie würde eine polyphyletische Gruppe aussehen?
Könnt ihr den jüngsten gemeinsamen Vorfahren (MRCA) von Adansonia suarezensis, A. rubrostipa und A. madagascariensis mit einem Punkt markieren? Wieviele Arten umfasst diese Klade?